
医学院 新闻动态
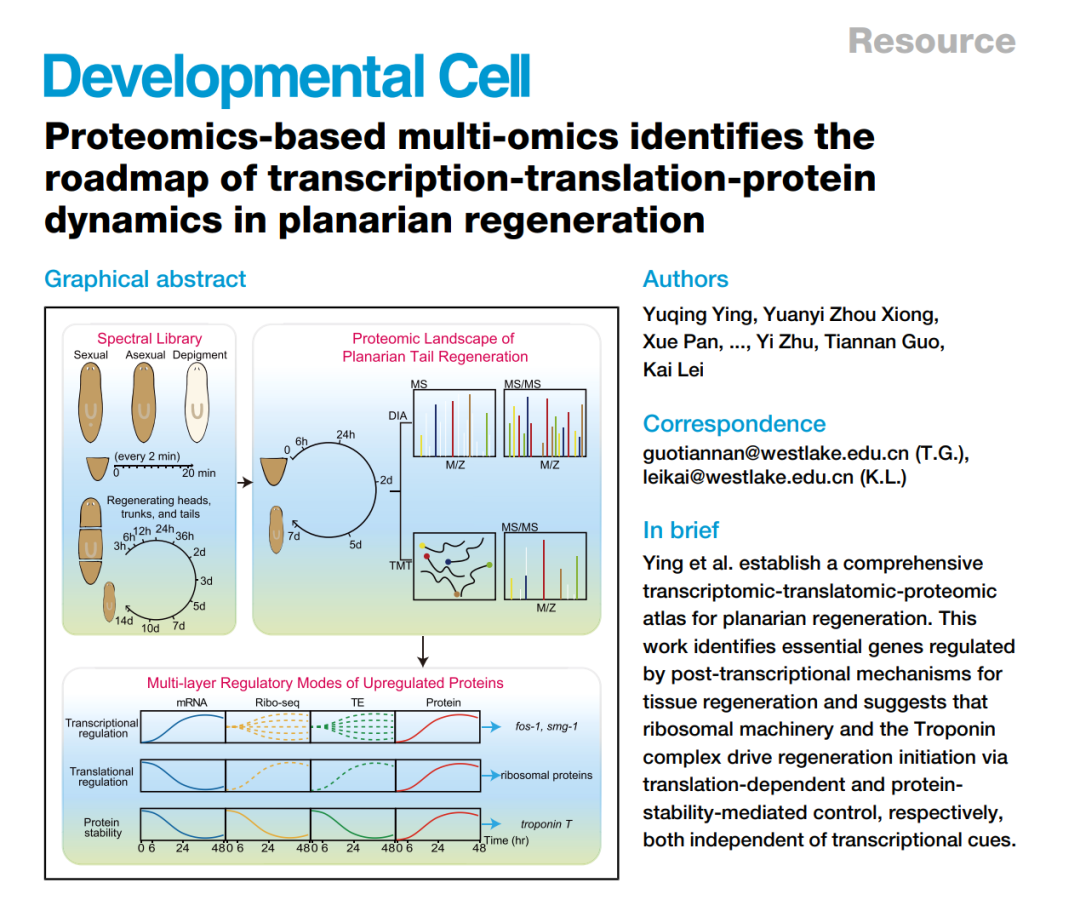
近日,西湖大学生命科学学院/西湖实验室雷凯团队与西湖大学医学院/西湖实验室郭天南团队合作在Developmental Cell和PNAS上分别发表题为“Proteomics-based multi-omics identifies the roadmap of transcription-translation protein dynamics in planarian regeneration”和“Pdgf mediates a transient regeneration-activated cell state in planarian tissue regeneration”的研究成果。
文章链接:https://www.cell.com/developmental-cell/fulltext/S1534-5807(25)00536-2
文章截图:
文章链接:https://www.pnas.org/doi/10.1073/pnas.2501874122
文章截图:

科学问题
如何驱动受损和衰老组织再生为功能完整的新组织,是生物学基础研究和临床医学转化领域长期面临的重大科学挑战之一。随着干细胞研究的突破,再生医学迅速发展,但体内组织器官“从无到有”的再生过程的调控机制,至今仍未明确,相关研究进展相对缓慢1。自然界中,涡虫、蚯蚓、蝾螈、斑马鱼等生物具备强大的再生能力,为解析再生机制提供了理想的研究模型2。目前已知,无论生物再生能力强弱,组织损伤后早期均会出现ROS水平升高、jun-1/fos-1基因转录激活、ERK磷酸化等损伤响应信号,但决定再生是否启动的后续关键调控环节与分子机制,仍存在大量未知,这也成为制约再生医学发展的核心瓶颈。
地中海圆头涡虫Schmidtea mediterranea(以下简称涡虫)作为三胚层扁虫的代表,拥有极为罕见的全个体再生能力2,3——即使被切割成约1mm的组织块,每个组织块仍能再生为功能完善的新个体4。这一特性依赖于干细胞增殖、伤口反应诱导、位置控制基因表达等多环节的精确协同。但此前对于这一过程的相关研究多聚焦于基因转录层面的动态变化,对转录后调控,尤其是蛋白质层面调控是如何驱动再生启动的认知仍较为匮乏,亟待系统性探索。
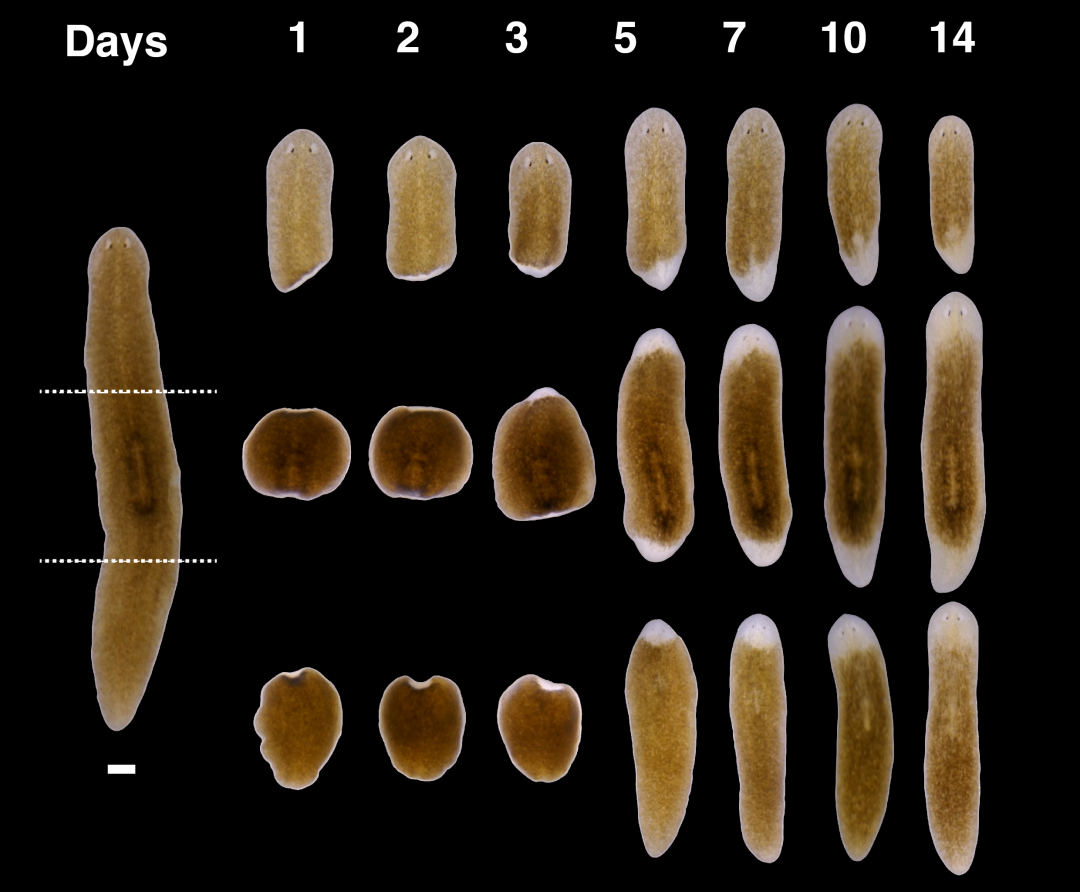
图1 涡虫再生过程
研究进展
雷凯团队长期聚焦再生机制研究,围绕“再生启动的分子调控网络”开展了一系列的系统性工作。在前期研究中,团队成员发现rRNA表观修饰可通过调控不同细胞内特定mRNA翻译,影响细胞再生命运(The EMBO Journal,2024)5。在此基础上,雷凯课题组联合郭天南课题组进一步对涡虫开展了基于质谱的蛋白质组学研究,构建了迄今最全面且可扩展的涡虫多肽谱文库(涵盖约10,000种蛋白质),并通过定量分析系统描绘了再生过程中蛋白质组的动态变化。
为精准识别再生启动关键蛋白,团队结合RNA-Seq(转录组)与Ribo-Seq(翻译组)数据,将蛋白质丰度升高分为“转录调控依赖”“翻译调控依赖”“蛋白质稳定性调控依赖”三类,并通过功能筛选鉴定出对再生必需的核心蛋白。研究揭示了一种核糖体介导非转录依赖调控机制,该机制在涡虫再生启动中发挥关键作用。研究还同时发现troponin T(肌钙蛋白 T)的蛋白质丰度会先于转录与翻译上调而升高,且这一过程依赖蛋白质稳定性调控,而非传统认知的转录激活。
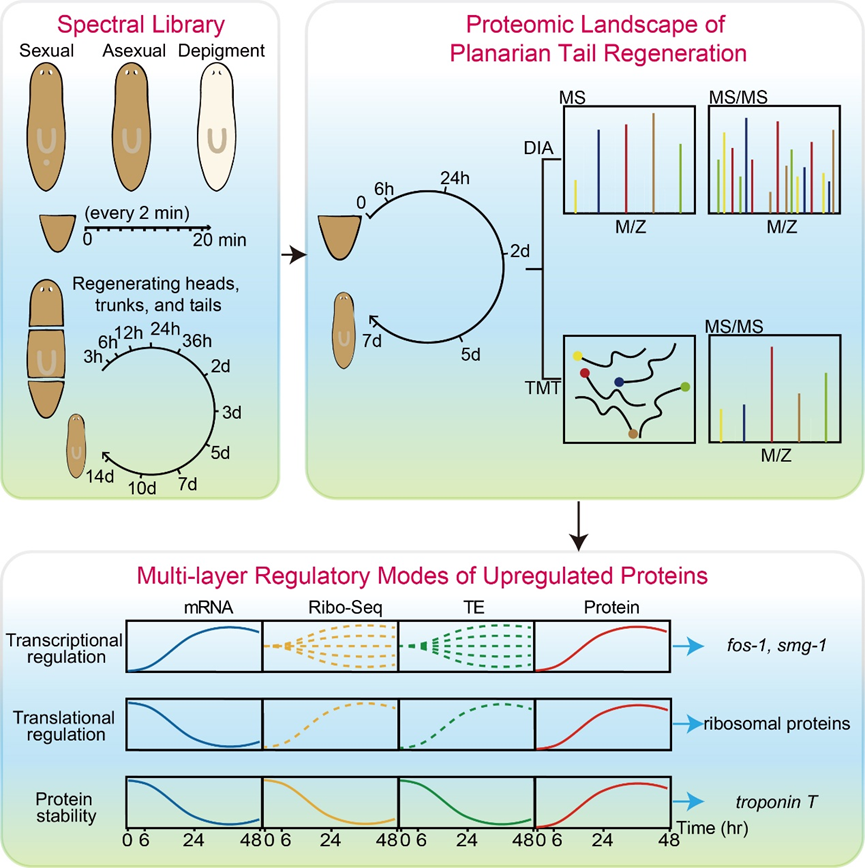
图2 涡虫再生过程的“转录组-翻译组-蛋白质组”多组学整合图谱构建和机制发现
以上研究基于中心法则,成功构建了涡虫再生过程的“转录组-翻译组-蛋白质组”多组学整合图谱,明确了核糖体亚基(翻译依赖)与肌钙蛋白复合物(蛋白质稳定性依赖)在再生启动中的核心作用,为再生调控的多维度系统网络分析奠定了关键基础。
在探索再生启动信号调控通路的过程中,雷凯团队此前已取得系列成果:2016年发现EGF信号可调控涡虫成体干细胞再生响应中的不对称细胞分裂(Developmental Cell, 2016)6,为理解涡虫成体干细胞如何精准修复损伤组织提供了理论模型;2021年在涡虫成体干细胞鉴定(Cell Regeneration, 2021)7过程中,团队成员发现了一个基因的表达模式呈现“瞬时再生激活细胞状态(transient regeneration-activated cell state,TRACS)”。基于这一观察,研究团队通过蛋白质结构域分析和AlphaFold2结构预测,鉴定这一基因是与人类PDGFB具有保守结构域和高度相似三维结构的涡虫PDGF同源基因,命名为Smed-pdgfl。研究发现,pdgfl的表达动态能够区分“缺失组织型损伤”(最终启动再生)与“非缺失组织型损伤”(仅启动伤口愈合),揭示了机体识别损伤类型的分子标志物。通过RNA干扰、荧光原位杂交、免疫染色等实验,团队进一步证实:缺失组织损伤后,pdgfl的表达会从“主要由肌肉细胞表达”动态转换为“干细胞表达”,约1周后恢复至肌肉细胞表达模式,完美匹配此前发现的“TRACS状态”8,且这一转换过程依赖ROS与pERK信号的短距离激活。功能验证显示,敲降pdgfl表达会导致肠道和中枢神经系统再生缺陷,同时引起前极标志基因notum和sfrp-1表达下降及肌肉前体细胞(myoD+PIWI-1+)积累,表明pdgfl不仅是损伤类型识别的关键信号,还在再生极性建立与肌肉细胞分化中发挥关键作用。
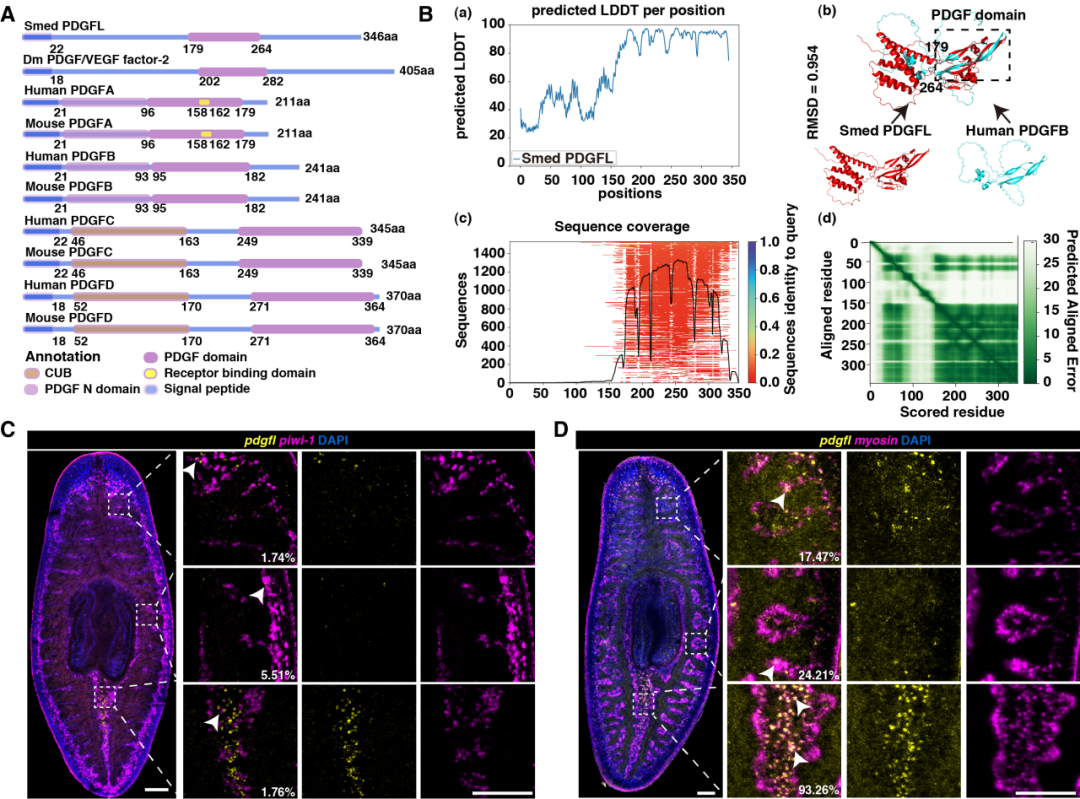
图3 地中海圆头涡虫中PDGF同源蛋白的鉴定
综上所述,这两项工作是雷凯课题组持续揭示“再生启动调控机制”的阶段性成果。课题组近期还揭示了线粒体形态动态对涡虫个体再生的调控功能与机制(Nature Communications,2024)9,提示线粒体正常稳态调节可能是决定再生是否启动的关键调控中心之一。这些发现进一步丰富了再生启动的调控网络,为多维度解析再生机制提供了新方向。雷凯课题组亟待在“再生启动调控机制”这一研究方向上持续获得新的发现。
在第一项研究中,西湖大学博士研究生应雨晴、熊周元一、潘雪,原西湖大学科研助理张秋实为本文共同第一作者。西湖实验室、西湖大学生命科学学院特聘研究员雷凯、西湖大学医学院特聘研究员郭天南为本文通讯作者,西湖大学博士研究生陈佳佳、赵芸,西湖大学博士后蔡雪,原西湖大学科研助理伊笑,西湖实验室研究员朱怡为本项工作提供了重要帮助。
在第二项研究中,西湖大学博士研究生徐昊为本文第一作者。西湖实验室、西湖大学生命科学学院特聘研究员雷凯为本文通讯作者,西湖大学博士研究生李玉聪,西湖大学高性能计算中心李南、杨岭为本项工作提供了重要帮助。
郭天南课题组(guomics.com)长期从事蛋白质组学相关研究,联合人工智能,解析生物过程的原理,助力疾病诊疗。团队诚邀有志于AIVC研究的优秀本科生、研究生及博士后研究人员加盟!
参考文献
1.Sánchez Alvarado, A., and Yamanaka, S. (2014). Rethinking differentiation: stem cells, regeneration, and plasticity. Cell 157, 110-119. 10.1016/j.cell.2014.02.041.
2.Sánchez Alvarado, A., and Tsonis, P.A. (2006). Bridging the regeneration gap: genetic insights from diverse animal models. Nat Rev Genet 7, 873-884. nrg1923 10.1038/nrg1923.
3.Birnbaum, K.D., and Sánchez Alvarado, A. (2008). Slicing across kingdoms: regeneration in plants and animals. Cell 132, 697-710. S0092-8674(08)00141-4 10.1016/j.cell.2008.01.040.
4.Morgan, T. (1900). Regeneration in planarians. Archiv. Entwick. Mech. 10, 58-119.
5.Chen, J., Li, Y., Wang, Y., Wang, H., Yang, J., Pan, X., Zhao, Y., Xu, H., Jiang, P., Qian, P., et al. (2024). Fibrillarin homologs regulate translation in divergent cell lineages during planarian homeostasis and regeneration. EMBO J 43, 6591-6625. 10.1038/s44318-024-00315-x.
6.Lei, K., Thi-Kim Vu, H., Mohan, R.D., McKinney, S.A., Seidel, C.W., Alexander, R., Gotting, K., Workman, J.L., and Sánchez Alvarado, A. (2016). Egf Signaling Directs Neoblast Repopulation by Regulating Asymmetric Cell Division in Planarians. Dev Cell 38, 413-429. 10.1016/j.devcel.2016.07.012.
7.Niu, K., Xu, H., Xiong, Y.Z., Zhao, Y., Gao, C., Seidel, C.W., Pan, X., Ying, Y., and Lei, K. (2021). Canonical and early lineage-specific stem cell types identified in planarian SirNeoblasts. Cell Regen 10, 15. 10.1186/s13619-021-00076-6.
8.Benham-Pyle, B.W., Brewster, C.E., Kent, A.M., Mann, F.G., Jr., Chen, S., Scott, A.R., Box, A.C., and Sánchez Alvarado, A. (2021). Identification of rare, transient post-mitotic cell states that are induced by injury and required for whole-body regeneration in Schmidtea mediterranea. Nat Cell Biol 23, 939-952. 10.1038/s41556-021-00734-6.
9.Pan, X., Zhao, Y., Li, Y., Chen, J., Zhang, W., Yang, L., Xiong, Y.Z., Ying, Y., Xu, H., Zhang, Y., et al. (2024). Mitochondrial dynamics govern whole-body regeneration through stem cell pluripotency and mitonuclear balance. Nat Commun 15, 10681. 10.1038/s41467-024-54720-1.

















